 点击JMOL窗口中的一个原子可以产生一个识别报告,如左下角的分子(见右边快照), 并且在浏览器的状态栏里。
(在
Firefox的Mac OSX版本 里, 需要点击两次才能产生报告)
氨基酸报告为3个字母的缩写,
核苷酸是 A, C, G, T, 或 U。
配体和非标准残基是其他缩写。
假如你不认识缩写,请点击PDB图标(在 FirstGlance 左上角窗口), 在那有生物学&化学标签,在下面可以看到配体和修复控件;或者点击OCA图标(在 FirstGlance 左上角窗口), 在下面有配体控件。
点击JMOL窗口中的一个原子可以产生一个识别报告,如左下角的分子(见右边快照), 并且在浏览器的状态栏里。
(在
Firefox的Mac OSX版本 里, 需要点击两次才能产生报告)
氨基酸报告为3个字母的缩写,
核苷酸是 A, C, G, T, 或 U。
配体和非标准残基是其他缩写。
假如你不认识缩写,请点击PDB图标(在 FirstGlance 左上角窗口), 在那有生物学&化学标签,在下面可以看到配体和修复控件;或者点击OCA图标(在 FirstGlance 左上角窗口), 在下面有配体控件。
原子确定使用PDB文件格式的名称,具体情况如下:
|
CA = 碳,α CB =碳,β CG, OG = 碳/氧,γ CD = 碳,δ NE, OE =氮/氧, ε NZ = 氮,ζ NH = 氮,η |
O3*, O5* = 3'和5'戊氧 C1* to C5* =1'和5'戊碳 C2-C8, N1-N9, O2-O6 = 基本元素 C5M = 五甲基碳(胸腺嘧啶) |
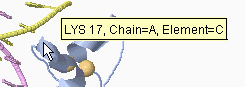 除了点击之外,你只要用鼠标简单的接触一个原子 (不需要移动鼠标,这是所谓的“徘徊”), 持续大约二秒钟后,在光标附近会出现一个黄色的原子识别报告(见右图)。这是在没有自旋的状况下。
除了点击之外,你只要用鼠标简单的接触一个原子 (不需要移动鼠标,这是所谓的“徘徊”), 持续大约二秒钟后,在光标附近会出现一个黄色的原子识别报告(见右图)。这是在没有自旋的状况下。
2006年1月,上面的悬停报告在 Mac OSX 10.3.9 版本下不会显示, 但是Mac OSX 10.4(" 老虎 ")版本下会显示 . 点击报告在两个版本中都会显示。
FirstGlance in jmol 完全Jmoljava小程序实现分子可视化。 为了能让Jmol正常工作,它需要一种高级程序叫java虚拟机。 为了能让Jmol正常工作,它需要一种高级程序叫java虚拟机。 计算机操作系统(如 Windows、 Mac OSX 和 linux)通常 包括java在内的,但是它可能是过时的版本。一般建议你及时跟新java ,跟新java可以访问java.com 。直到你访问 java.com。有些windows 电脑根本就没有java。
这是20种标准氨基酸,及其3字母和一字母缩写。 助记符可以帮你记住这些缩写,以不至于弄错。
|
Ala A 丙氨酸
Arg R 精氨酸 Asn N 天冬酰胺 Asp D 天门冬氨酸 (助记符:asparDic ) Cys C半胱胺酸 |
Gln Q 谷氨酰胺 (助记符:Quetamine) Glu E 谷氨酸 (助记符:gluEtamic) Gly G 甘氨酸 His H 组氨酸 Ile I 异亮氨酸 |
Leu L 亮氨酸
Lys K 赖氨酸 (助记符: liKesine) Met M 甲硫氨酸 Phe F 本丙氨酸 (助记符: Fenylalanine) Pro P 脯氨酸 |
Ser S 丝氨酸
Thr T 苏氨酸 Trp W 色氨酸 (助记符: tWyptophan) Tyr Y 酪胺酸 Val V 缬氨酸 |
用下面的方法查看氨基酸和核苷酸序列:
- PDB按钮,在右上方附近,有序列详述选显卡。有 单字母序列注释连接到其他资源。
- OCA按钮,在序列源信息选项下,单字母FASTA格式序列连接到其他资源。
- S2C可以通过OCA按钮。在OCA,使用SEQRES到坐标。
空缺: S2C(SEQRES坐标)对3D结构数据是非常有用的,但目前没有核酸资料)它列出的在蛋白质结构预测实验中的蛋白质序列符合给出的氨基酸坐标。 通常情况下,在一些晶体实验中,一些氨基酸无序晶体,并没有原子坐标。这些数据的空缺,红色部分无数据, 2ACE分子S2C解析中的摘录.
- S列是蛋白质实验中的序列; (从PDB文件头中的SEQRES 记录)
- C列是带坐标的氨基酸序列。 (从PDB文件中的原子记录)
***** SAMPLE FOR 2ACE *****
S C S C
SEQCRD 0 D ASP --- 1 - - - 367 3字母编码
SEQCRD 0 D ASP --- 2 - - - 367
SEQCRD 0 H HIS --- 3 - - - 367 这不是你的分子
SEQCRD 0 S SER SER 4 4 C T 5 2ACE分子作为列子。
SEQCRD 0 E GLU GLU 5 5 C T 5 为你的分子,
[lines 6-482 omitted] SEQCRD 0 N ASN ASN 483 483 C T 5
SEQCRD 0 E GLU GLU 484 484 C T 5
SEQCRD 0 P PRO --- 485 - - - 367
SEQCRD 0 H HIS --- 486 - - - 367
SEQCRD 0 S SER --- 487 - - - 367
SEQCRD 0 Q GLN --- 488 - - - 367
SEQCRD 0 E GLU --- 489 - - - 367
SEQCRD 0 S SER SER 490 490 C T 5
SEQCRD 0 K LYS LYS 491 491 C T 5
[lines 492-533 omitted]
SEQCRD 0 A ALA ALA 534 534 H H -
SEQCRD 0 T THR THR 535 535 H C 5
SEQCRD 0 A ALA --- 536 - - - 367
SEQCRD 0 C CYS --- 537 - - - 367
表中显示出缺乏1-3,485-489 、和 536-537 坐标。在S2C表中像上面的空缺很多,你可以用你的浏览器搜索栏在网页上输入关键词“---”查看到任何缺口。。
在FirstGlance查看 2ACE ,点击 N->C 彩虹, 然后粘贴
4, 484, 490, 535到查找并按ENTER键。你将会在链上看到用光圈标记的缺口。通过点击链(标有光圈)的开始或结束你会看到,链不是从1开始也不是到537结束。请注意,在没有审查的S2C表中,容易忽视晶体模型中的失踪残基
It is envisioned that a future version of FirstGlance 这是设想,未来版本的FirstGlance可能有一个内置序列表,序列表包含的信息来自S2C。点击设想清单中的残基可以在3D分子图形中加亮。规格已经指点好了,当实现并不容易。欢迎有兴趣的义务工程师联系
感谢王国立和Roland L. Dunbrack提共的S2C服务,并联系Jaime Prilusky安装上述的S2C服务。
- 亚基1没有(蛋白质、核酸,水), 那些被成为Jmol术语 " 蛋白质 "的蛋白质, 20 标准的氨基酸 (Jmol 有时候无法识别当做蛋白质的), 不包括钙 (其中不包括Jmol" 蛋白质 " ) [更多.]
这个亚基包括基体或抑制剂, prosthetic 如NAD的辅因子,碳水化合物,金属离子,等等,不管这些是共价和非共价键结合蛋白质或核酸链。 例子是1D66中的镉, 2HHD中血红蛋白(HEM), 1IGT中糖基化的蛋白1IGT,和 1APM中洗涤剂(OCT)和磷酸化(PHO)的丝氨酸和苏氨酸 。
很少,一个配体可能是无法显示, 但可以用非标准残基非标准残基代替。一个列子是 1JSA中的1JSA。JMOL混淆GLY1都不插入代码(见Jmol缺陷).
- 亚基2包括不属于聚合链的标准氨基酸和核苷酸(单一的残基,并非链中的残基)或那些缺乏一定标准原子的氨基酸和核苷酸。假如不包括配体,拿在卡通,二级结构, N->C 彩虹,和vines显示模式中配体被隐藏。.[更多..]
列子包括在 4CPA中GLY1:G,和在1BKX中A351:B.
在极少数情况下,能识别共价键(见Jmol缺陷)。这可能导致一个残基不是会出现在链上。 例如 2SOCJmol不中的THR6,又见异常原子。 又见 异常原子。
Jmol有DNA and RNA 定义。这些定义还没有发现错误
蛋白质 在Jmlo里被定义,但定义是不严谨的 (在 10.00.48版本) 。值得注意的是,钙也包括在内,并且很少有标准氨基酸不包括在内(例子)(例子). 因此, FirstGlance定义蛋白为:
使用Jmol定义的蛋白包括一下非标准氨基酸,意义(见下节)
FirstGlance里的碳水化合物定义如下:
以上的名单是不可或缺的。因为,在Jmol定义里他们不像不像 " 蛋白质 " , " DNA", 和 " RNA", " 碳水化合物 "中的一种 。)蛋白质数据库可以验证上表中的每一组。上表的汇编有几个来源,包括与Thomas Luetteke(麻省理工学院 Thomas_L)的讨论, 并在数据库中被Jaime Prilusky核实,SWEET2中有模板。 表不包括硫酸盐和少于每10个PDB条目中出现的糖基化糖。假如你有关于上表的任何改进建议,请发送到(附带PDB编号)
初始化,非标准残基被显示为十字架::

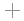
十字架能被藏着,或非标准残基被显示为球和棒状,使用控件可以浏览 更多效果...
定义.蛋白质数据库格式文件(PDB文件)定义了标准氨基酸和核苷酸, 并且有原子记录类型。 这些包括:
- 20 标准的含氨基的酸,以及模糊的残基密码,ASX,GLX,和未确定的UNK.
- 六种标准核苷酸 A, C, G, I, T, U附带 UNK。( PDB格式文件没有区别 ribo- 和deoxyribonucleotides残基密码。)
- 修正标准的核苷酸指定为+A, +C, +G, +I, +T, +U.
脊椎追踪。非标准氨基酸和核苷,或非标准的原子被记录为HETATM类型。不管带有原子残基或HETATM残基的标准主链,Jmol通常认为这些残基"蛋白质", "DNA", or "RNA"都包括在脊椎追踪 (一种不似乐钟的丰富特征)。
区别非标准残基。 当 Jmol in FirstGlance 被要求突出非标准残基时(配体或跟多问题的帮助复选框), 它这样做选择" hetero 和 (蛋白质,核酸) " 。
- 只有非标准残基中的原子(主要包括标准主连原子)被认为是PDB文件中的HETATM类型时,这种方法才是正确的。例子包括1EVV(2MG, H2U, M2G, OMC, OMG, YG, PSU, 5MC, 5MU, 7MG, and 1MA)t-RNA中的许多非标准核酸,1H3O中的非标准seleno-methionines,和1BKX中的标准磷酸化serines和threonines。一般的,在这些情况,整个非标准HETATM残基会给予一个PDB残基密码名,如selenomethionine命为MSE, phosphoserine命为SEP 、或 phosphothreonine为TPO。 对应的全称在PDB头文件的MDRES和HETNAM文件中(可从 pdb.org查看,如Header
of 1EVV), 或点击FirstGlance控制选款(左上角)的PDB或OCA连接。
- 当PDB文件中使用选择协定时,这种方法是不正确的:是指带有非标准残基的标准主链原子,并且只是HEATM类型的非标准原子。如1B07中的带有PYL1005的GLY5,1APM中的磷酸丝氨酸(带有磷酸基的丝氨酸)和磷酸苏氨酸(带有磷酸基的苏氨酸),310D中的甲基 ribonucleotides(例如甲基+C1)。这是不能更改的,在这种情况下,FirstGlance显示非标转蛋白的残基为“配体+”。
- 绝少,非含氨基的酸会显示为非标准氨基酸,举如 1JSA中的MYR1。纠正这类问题正在审议中。 More..
如果你们知道如果你知道上述没有提到过的起错误,请发送PDB简码到
你可以输入任何的原子表达,从Jmol的command language,进入查找栏,例如:
- RNA
- MSE (selenomethionine的组名,如1K28)
- 12-23 (所有残基物/范围内的序列组)
- 12-23:B (残基/B链范围内的序列组)
请注意, " 晕轮原子 "(从查找栏..) 可以指定为目录的标签。。显示. (见为目的的联系复选框..) 那是,你能直观的看到非共价相互作用的原子与“晕轮原子”因此,查找..可以用来选取目标有问题的选择与控制选择目的在联系.. 界面
- " 晕轮原子 " 是什么? 使用查找..
左上角有晕轮云子连接。 晕轮是亮黄色的. (见右边的快照--鼠标移到快照你能看到更多信息)
- Why aren't all residues in the chain selected?为什么链中的残基不能全部被选定? 当你点击一条链时,非标准残基(如果有的话)不能被选定-只有标准残基能被选定。(非标准残基染色比标准残基更深。但同样色调的其余链。你能在多种浏览模式下是哦那个复选框来加亮非标准残基或者配体。)这样做的好处是,你能看到标准残基(目标)与非标准残基之间的联系。加入你想添加标准残基目标的话,有两种方法。
- 如果只有少数几个,残基/组选项模式来改变并且点击他们中的每个。
- 如果有很多,使用查找。晕轮周围请求目标。例如你输入特定的链(带有冒号的前缀名字,例如:A为链A的意思),或蛋白,DNA,或RNA。(几日非标准氨基酸有晕轮)然后联系..并且检查晕轮原子。下一步,参看原子联系对面。在这点上,你可以点击查找..并且确认晕轮,然后返回联系(使用左下角上部的联系返回连接)
- 如果只有少数几个,残基/组选项模式来改变并且点击他们中的每个。
- 为什么我不能通过点击其他来取消一选择项?为什么我不能通过点击其他来取消一选择项?一般的,当你选
择了点击它,你可以再次点击取消他。例如,如果你选择模式
是“链”,你已经选择了A链,再次点击A链可以取消。然而,
当你点击的东西,包含一个更大的原子选项组,那不会取消。例如:
- 假如C链在选定状态,并且更改选项模式为“残基/组”点击C链的残基,残基不会取消。解决方法:要选取部分大型链,使用“一条链中的范围残基”模式。
- 如果" 晕轮原子 "在选定状态,而且选项模式是 "残基/组 ", 你不能通过点击其他原子来取消晕轮原子选定。 解决方法: 不检查“晕轮原子”。 现在,在 " 残基/组 "选型模式下,使用晕轮作为知道点击你想要的晕轮组。
- 假如C链在选定状态,并且更改选项模式为“残基/组”点击C链的残基,残基不会取消。解决方法:要选取部分大型链,使用“一条链中的范围残基”模式。
- 如何使用残基范围内的序列号?假设你以22-23号序列为目的。在搜索栏..输入“22-23”,22到23号12个残基放入晕轮。现在点击联系..目前,检查目标晕轮原子。现在你以准备好了原子联系目标。如果有多于一个的链有22-23好残基,在你想要的链欠佳益而高冒号。如链B,输入22-23:B在查找...


该机制是用来选择匿名链的纤细 技术信息.
FirstGlane联系.. 栏显示你选在的任何一类目标非共价(共价)原子。像这类原子被认为是“联系”,并且被显示为球形(无论是对范德华半径,或者跟小的半径在“球棒”模型)。“联系”原子完全取决与与目标原子的距离,才用下面的标准。欢迎对标准提供改善建议(见页面底部的反馈地址)。
Jmol 有显示DNA碱基之间的氢键的内置机制,并且在蛋白质的二级结构骨架中。这一机制是最基本的,因为实际上现在它只能显示氢键的一小部分,比且他没有被FirstGlance使用。(详述,氢参阅Jmol Reference Manual。) FirstGlance只显示近端原子对,并把他们留个阅者来判断他们之间是否为非共价键。
- 假定氢键:氢和氧原子距离是是3.5 Å 内。蛋白质的氢键和供给受体的距离小于3.2Å是有重要意义的。当供给少提的距离超过3.5Å键能是无意义的。
更多关于氢键。当一对供给受体氢键间的距离在3.5Å内时,供体和受体间会有氢键,并且供给质子与受体电子对之间有一定的角度。氢原子位置的检测是非常有用的。然而,似乎强力重大氢键的出现不符合角度标准
(Morozov et al., 2004; 见以下例子)。- 氢键的简单例子:1D66 蛋白链识别DNA序列CGG通过氢键。
-
缺少的氢键可以通过MolProbity服务器加到PDB文件里。(见更多浏览..的下的连接,全部原子联系分析)保存结果模式到磁盘后,可以在firstglance.jmol.org上传测试 。谨慎起见,然而,供给受体距离和角度的准确性依靠分析方法和当时的测定环境(在更多浏览..下见质量模式)
- 非键的例子:Nelson et al. (2005) 的在“干燥表面“的淀粉类纤维结构报告出现的氢键。( 见Jmol三维图片)被确定不是严格角度测试的结果。(David Eisenberg,私人交际)
- 氢键的简单例子:1D66 蛋白链识别DNA序列CGG通过氢键。
- 水和水桥(Water birdges)。
在关联显示,能区分一带水的氢键和两者都不是谁的氢键。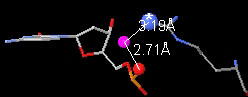 1D66侧链精氨酸氧46(B链)与胞嘧啶13(链D)中磷酸之间的水桥
1D66侧链精氨酸氧46(B链)与胞嘧啶13(链D)中磷酸之间的水桥
此外,水桥可以单独显示(包括水的氢键的一个亚基)每个水桥包括至少下面三类原子: - 范德华力。4.5 Å内的碳、硫或硒。这些足够密产生显著的范德华“疏水性”力 ,包括C-S, C-Se, 和S-S相互作用(包括目标和关联目标间的二硫键,例如,抗体1IGT中的两条重链)
- 疏水作用的简单例子:1D66的两条链相互接触形成一个小的,隐藏的,疏水核心。
- 盐桥:中性PH值内带正点的侧链氮(arg.ne,arg.nh?,lys.nz)和带负电的侧链氧(asp.od?,glu.oe?)间的距离在4.0 Å内。
更多关于盐桥..
FirstGlance 中有两种方法浏览盐桥:(i) 联系.. 显示盐桥是你的目标选项和剩余部分的模式; (ii) 更多视野..使你能看到它们全部的模式 。
- 例如: 在 2HHD 里在A链和C链间每个方向都有一个盐桥。
- pi轨道阳离子的相互作用:
氮阳离子(赖氨酸和精氨酸)面对芳香环在6.0A内时,产生的积极的相互作用力被叫做“pi轨道阳离子的相互作用”。(Gallivan
& Dougherty, 1999)有一个简单的介绍,看
更多关于pi轨道阳离子的相互作用
。
有两种方法在 FirstGlance中查看pi轨道阳离子的相互: (i)联系.. 显示它们是你的目标选项和剩余部分的模式 (ii) 更多视野..使你能看到它们全部的模式。- FirstGlance 显示以可能的PI阳离子相互作用 以阳离子与3个间隔碳的芳香环6.0A内为基础。FirstGlance 无法显示极性大的PI阳离子的相互作用,和一些表明极性大的。请查看一个关于任何PDB文件PI阳离子相互作用可靠列表的 CaPTURE 服务器。
有时候, FirstGlance 会显示没有接触环的赖氨酸和精氨酸。这是一个很难修正的缺陷。FirstGlance的目前规定是寻找3环原子在 6.0 Å内,但是无法判断这三个原子在同一环内。一个列子是 2HHD,它的链A和C中的ARG141显示是没有环伴的。这是因为TYR35和TRP37都有 6.0 Å内的环原子,但是没有一个环用这样的三个原子。CaPTURE认定TRP37的相互作用极性很大,尽管他没有违反 6.0 Å规则。
- FirstGlance和CaPTUR都不会报告没有标准氨基酸组成的阳离子和芳香环的PI阳离子相互作用。例子包括金属阳离子,或芳香环配体。FirstGlance能,不过可以显示任何关联成分,所以你可以选择金属或配体芳香环,并且显现他们的接触。Protein Explorer是提供给显现这些的高级工具。
-
例子:
- 2VAB的缩氨酸链P的一端包含 PHE1:P并互相影响,以根据CaPTURE的一个极其重要的方式,与LYS66:A。
- 同源四聚体1BL8中的每两条链之间都有PI阳离子相互作用。有些被CaPTURE认为是极其重要的。
- 1B07中的缩氨酸链C与链A有极其重要的PI阳离子作用力 。
- Firstglance 发现两个赖氨酸:在1IGT中的链B和D,每个都有PHEPi阳离子相互作用。CaPTURE无法处理PDB文件包含“插入”序列号,这包括 1IGT。但是,加入PDB文件被重新编号(带有RasMol)和保存,然后上传到CaPTURE,其结果就不一样了。
CaPTURE不会让任何东西别FristGlance接近察觉认为是极其重要的。
- 金属与各类键包括三类距离基础的原子对。
- 硫原子与一个金属原子(假定是一个阳离子)在3.2 Å。" 金属 " 被定义在2006年4月在蛋白质数据库中发出现的任何的金属,包括下列各项:
- 铋,钫
- 碱土:镭
- 过渡金属:钪,钛,铌,铪
- 稀土:铪,钕, promethium ,镝,铥
- 锕:锕,钍,镤,镎,钚,镅
- 铋,钫
- 不是目标的任何外部原子(碳,硫,磷或氢)和不是目标的任何原子(碳,硫,或氢)在3.5A内 ;反之亦然。原子被认为有氢键(见上文) ,然后在排除。由此产生的结果,例如,氧型金属,硒金属,金属与金属的相互作用,以及氯离子与极性原子的相互作用,但不包括金属离子与硫的相互作用。
- 不是目标的任何外部原子(碳,硫,磷或氢)和任何目标金属原子在
3.5 Å内 ;反之亦然。 发现像氢,氧这类原子与金属形成键,不论他们是否参与形成氢键。由此产生的结构,例如, 组氨酸侧链氮与血红铁离子形成的键。
- 不是目标的任何外部原子(碳,硫,氮,硫,磷,或氢)和任何目标原子在 4.5 Å 内 ;反之亦然。 这包括,例如,金属或氯离子与任何元素的相互作用、和 Se-C 或 Se-S 的相互作用。这项规则的目的在于没有预设前提规则的情况下, 显示成分见的相互作用。
- 硫原子与一个金属原子(假定是一个阳离子)在3.2 Å。" 金属 " 被定义在2006年4月在蛋白质数据库中发出现的任何的金属,包括下列各项:
在晶体模型中往往没有氢,因为X射线衍射通常不能测出氢。与此相反,目前的氢通常是核磁共振结果。在某些晶体模型,添加了氢的分子模型,要么只有极性原子,或所有原子。下面是 更多关于氢 。
- 氢在关联中显示:在”联系显示“展览中,只有那些接触原子(球)氢键被显示。(氢键在没有接触原子时,[棒]是不会显示的 )用棒模型显示氢与球的连接,这是的键角更清晰。
- 例如:选择 1LCD 单一的钠离子为目标,并查看其联系。水氢联系钠离子原理它,符合金属离子结合孤对电子
每个含有联系原子的氨基酸和核酸(包括非标准残基)显示标签。 (联系配体和溶剂不显示标签,点击这些识别他们) 标签格式
定义。 当原子不被Jmol认为是下列中的一个: 蛋白质,核的酸,或 " hetero".
根据 PDB 文件的规定,所有不是蛋白质和核酸标准残基成员的原子都应该被指定为 " hetero" 。 Hetero 原子通常的进一步细分: 有 " 溶剂 (水和一些像硫酸盐和磷酸盐的无机离子),其他都是“配体” 。 少有PDB 文件不遵从规则,并使Jmol出错。而且很少有, Jmol不正确地解释 PDB 文件。 结果是是既非蛋白质的,核酸,也不是hetero的原子。 在 FirstGlance 里面,这丢都被认为是反常原子。
 问题。 FirstGlance 无法正常显示异常原子。 有些异常原子除了在Vines模式下的其他浏览模式下都是不可见(详述)
问题。 FirstGlance 无法正常显示异常原子。 有些异常原子除了在Vines模式下的其他浏览模式下都是不可见(详述)解决方法。为了要避免留下异常原子不可见。他先被被显示为范德华辐射圆点(见右边快照)。这些圆点的颜色依靠视图。这些圆点容易与光晕区分,因为他们先对分散,且被安排在球面圆内而非平面的,并且通常是黄色的。为了识别反常原子,在更多视野..,你能够看到的球体部分。你可以点击他们查看。
更多视野。。有隐藏异常原子圆点的选项
例子:
-
缺失主链原子,这可能是缺乏电子密度,或是简单的因为错误的过期PDB文件。
- 1B07中的GLY5中的, 1UC5中MET79:M缺少主链氧(.o)。
- THR918 in
1H3O,
和
ALA333:C in
1PRC
除了缩氨酸氮键缺少所有原子。
PDB文件格式错误 - FOR0:H in
1PRC
应该是HETATM却是原子。
错误也可能源自Jmol编译 (版本 10.2.0). (见 Jmol缺陷,主链原子没有出现) - 1GRH只有二个残基、 CYS(原子)和 HEC(HETATM)。Jmol 无法识别 CYS当做蛋白质,而且无法连接它到主要链原子。
- 一些包含D氨基酸的录入 ,Jmol 无法认识一些标准 L 氨基酸当做蛋白质:
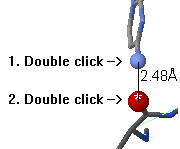 测量两个原子间的距离,双击第一个原子。现在你点击另一个原子,距离将被显示为红线相连原子(除了在Mac Firefox)。双击第二个原子距离报告。
测量两个原子间的距离,双击第一个原子。现在你点击另一个原子,距离将被显示为红线相连原子(除了在Mac Firefox)。双击第二个原子距离报告。如果你想取消距离修改报告,移动你的鼠标到Jmol工作页面。如果你想移动报,再次双击两个原子中的一个。如果你想移动所有的距离和角度报告,点击隐藏连接(在联系栏),或点击更多视图..然后隐藏距离和角度。(Jmol命令是“monitors off”。)
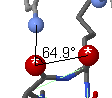 测量角度,双击第一个原子,然后单击第二个。在你接触额外的原子是,角度将会被以红线的方式显示。双击第三个原子产生角度报告。
测量角度,双击第一个原子,然后单击第二个。在你接触额外的原子是,角度将会被以红线的方式显示。双击第三个原子产生角度报告。 测量一个扭曲的(二面角)角度,同样的方法,除了需要第四个原子。
测量一个扭曲的(二面角)角度,同样的方法,除了需要第四个原子。
火狐 (getfirefox.com)是一个很好的,流行的,免费的,开放代码网页浏览器,在 Gecko家族。
Windows: 在windows操作系统,FirstGlance in Jmol在火狐1.0和1.5可以正常工作。 Firefox (和其他浏览器) 没有请求java升级的特殊浏览器, 但使用system-wide java.
Mac OSX: 在Macs操作系统Safari比Firefox合适, 即使 Firefox 1.5是开放的。 在点击识别原子或录入时,那是不完美的,通常需要点击两次纠正请求。同样的, 在距离和角度测量时,在接触最后一个原子后,结果可能不被显示;或即使显示了,在鼠标离开Jmol浏览栏时,线不会取消。 不管怎样,在双击后一个原子后结果会显示出来。
linux: 在Knoppix,火狐支持FirstGlance in Jmol,但我们没有linux下的充分信息。
又见 Safari 和 类似浏览器。
Safari 是苹果Macintosh OSX电脑操作系统的一款网页浏览器并支持FirstGlance in Jmol正常工作。然而 2005年以后, 在使用 Safari OS 10.3.9以后版本时,出现了一下问题: 有时连接和控制按钮不能正常的工作。简单的 退出和重启Safari可以解决,或升级到 OSX 10.4 Tiger来避免此类问题。
-
在同样的Safari session下,在访问FirstGlance in Jmol前访问其他的使用JAVA语言的网站时,出现问题。这个问题的出现源自在苹果中的java漏洞。这已经给他们发送了报告。
另一个类似异常是在Safari in OS 10.3.9 中的 "hover" 原子报告没有出现。 (在 OS 10.4中有)然而, 在所有的 Safari/OSX版本中,点击原子产生的报告在浏览页面的做下角。
又见 Mac Firefox 和 类似浏览器。
安全. 你的数据(必须是PDB格式,可以有其他后缀的文件)被加密传送,并暂存到位于以色列雷霍沃特魏茨曼科学研究所的生物信息和生物计算服务器上。然后,通过Internet重新传送到你浏览器的Jmol程序。电脑黑客可能会截取你的文件。机构需要有一个安全的方法来上传你的数据文件并显示在你的安装的FirstGlance in Jmol 里。有任何问题请随时联系
方法. Jaime Prilusky热心提供的CGI程序接受上传PDB文件。 你可以在你的上传栏里插入下面的HTML代码:
<form method='post' enctype='multipart/form-data'
action='http://bip.weizmann.ac.il/oca-bin/fgij'>
Upload a PDB file of your own:
<br><small>
Caution: uploading a PDB file may <b>disclose your data</b> to others.
</small><br>
<input type='file' name='file' size='50'>
<input type='submit' value='View in FirstGlance'>
</form>